Las “salas blancas” (cleanrooms) donde se ensamblan y prueban naves espaciales están diseñadas para mantener niveles extremadamente bajos de partículas y microorganismos. No es un capricho: si una sonda lleva microbios terrestres a otro mundo, puede comprometer experimentos de búsqueda de vida y, en el peor de los casos, introducir contaminación biológica no deseada.
En ese contexto, un equipo internacional con participación del Jet Propulsion Laboratory (JPL) analizó una colección histórica de bacterias aisladas durante la preparación de la misión Phoenix (2007) y, mediante secuenciación genómica, identificó 26 especies previamente no descritas entre los microorganismos presentes en esas instalaciones ultra-controladas. El trabajo se publicó en la revista Microbiome el 12 de mayo de 2025.
Visto desde fuera parece paradójico: ¿cómo puede haber biodiversidad nueva en un entorno que se limpia de forma agresiva? La respuesta corta es que estos lugares no están “vacíos”, sino que funcionan como un filtro evolutivo muy intenso: sobrevive lo que tolera desecación, escasez de nutrientes, radiación y químicos de limpieza.
Qué encontraron: 215 aislados, 53 cepas “candidatas” y 26 especies nuevas
El estudio parte de 215 cepas bacterianas cultivadas a partir de muestras tomadas en la Payload Hazardous Servicing Facility (PHSF) del Kennedy Space Center (KSC), asociada a la campaña de integración y pruebas de Phoenix. Los autores secuenciaron los genomas de los 215 aislados y concluyeron que 53 cepas correspondían a 26 especies nuevas (no descritas previamente) repartidas en 18 géneros.
Algunos datos técnicos relevantes que ayudan a dimensionar el hallazgo:
- Aproximadamente un 25% de los aislados (53/215) caen en ese conjunto de especies nuevas.
- En el conjunto aparecen miembros de varios grandes grupos bacterianos (por ejemplo, Bacilli y Proteobacteria), con representación también de actinobacterias.
- Muchas de estas cepas se aislaron tras someter las muestras a condiciones “selectivas” (p. ej., pH alcalino, choque térmico, baja temperatura, exposición UVC), lo que favorece la recuperación de microorganismos especialmente tolerantes.
Además, los autores intentaron responder a una pregunta crítica para la protección planetaria: ¿son frecuentes o residuales? Para ello mapearon lecturas metagenómicas de distintas salas blancas y observaron que las lecturas asociadas a estas especies nuevas eran muy raras (menos del 0,1%), lo que sugiere baja abundancia.
Cómo se descubren “especies nuevas” hoy: del cultivo a la genómica comparada
Hace años, identificar especies bacterianas nuevas dependía mucho de rasgos fenotípicos y de marcadores como el 16S rRNA. En este trabajo el núcleo es la genómica completa: secuenciación (incluyendo ensamblados de alta calidad), comparación de genomas y análisis filogenómicos para delimitar clados y distancias genéticas consistentes con especies distintas.
En términos divulgativos, el proceso puede resumirse así:
- Aislar y cultivar bacterias de muestras del entorno (superficies/suelos del cleanroom).
- Secuenciar genomas de forma masiva.
- Comparar genomas con bases de datos y calcular índices de “relación genómica” para ver si encajan en especies conocidas o si se separan de ellas.
- Validar con filogenómica y, cuando procede, con caracterización adicional.
Este enfoque es especialmente potente en salas blancas porque, al ser entornos oligotróficos (muy pobres en nutrientes) y muy estresantes, tienden a seleccionar microbios con rasgos raros que en otros ambientes quedan “diluidos” en comunidades más complejas.
Por qué sobreviven en salas blancas: esporas, biofilms y reparación del ADN
No es que estos microbios “ganen” siempre a los protocolos de limpieza, pero sí muestran señales genómicas coherentes con estrategias de persistencia:
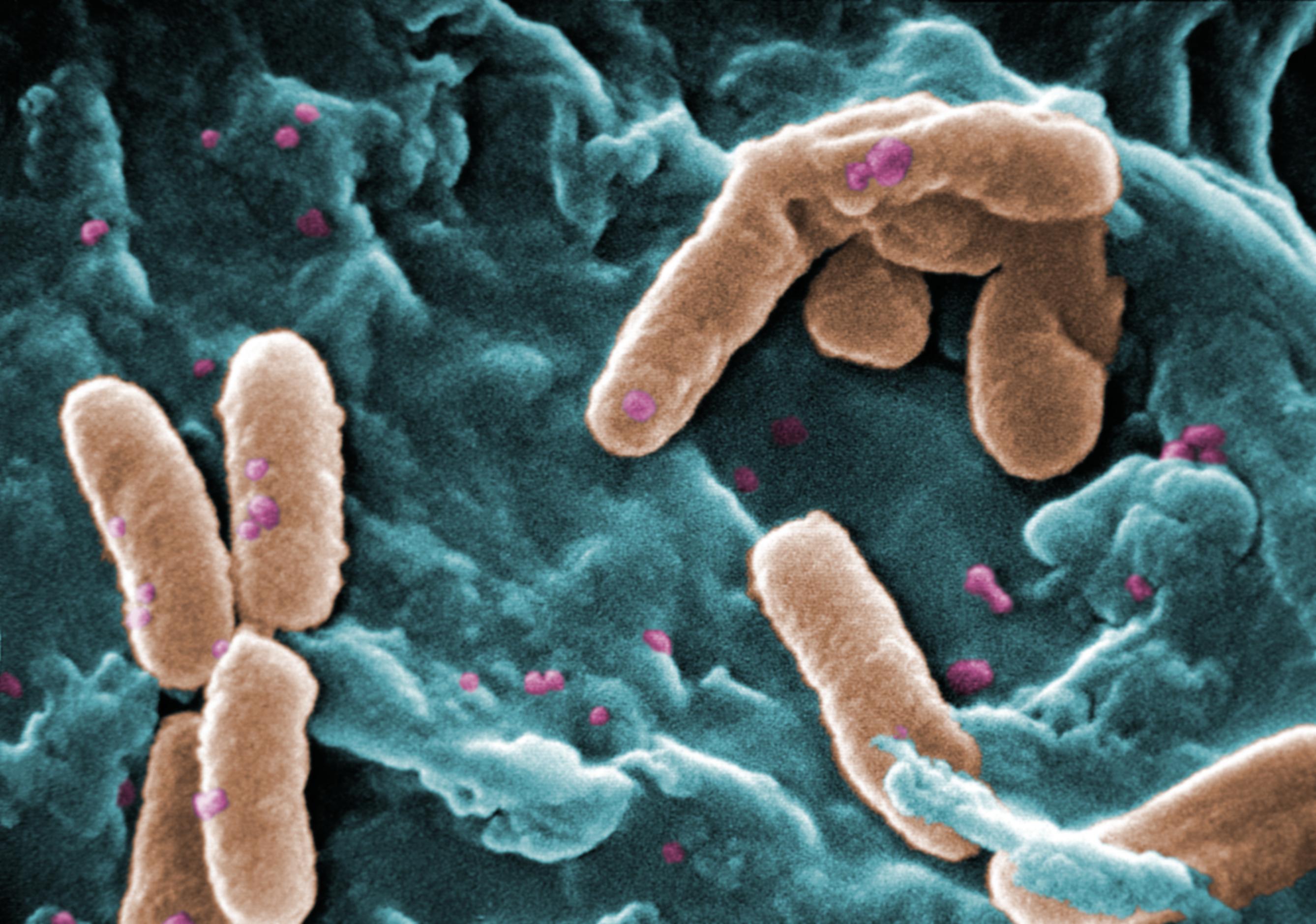 Crédito: CDC/Janice Haney Carr (PHIL), dominio público
Crédito: CDC/Janice Haney Carr (PHIL), dominio público
- Formación de biofilm (una matriz pegajosa que protege y facilita la adhesión), con genes asociados presentes sobre todo en algunos miembros proteobacterianos.
- Esporulación y reguladores del destino celular en especies formadoras de esporas, que les permite aguantar periodos prolongados en condiciones adversas.
- Proteínas y funciones vinculadas a resistencia a radiación y reparación del ADN / respuesta a estrés, detectadas en varios grupos, especialmente en esporulados y algunas actinobacterias.
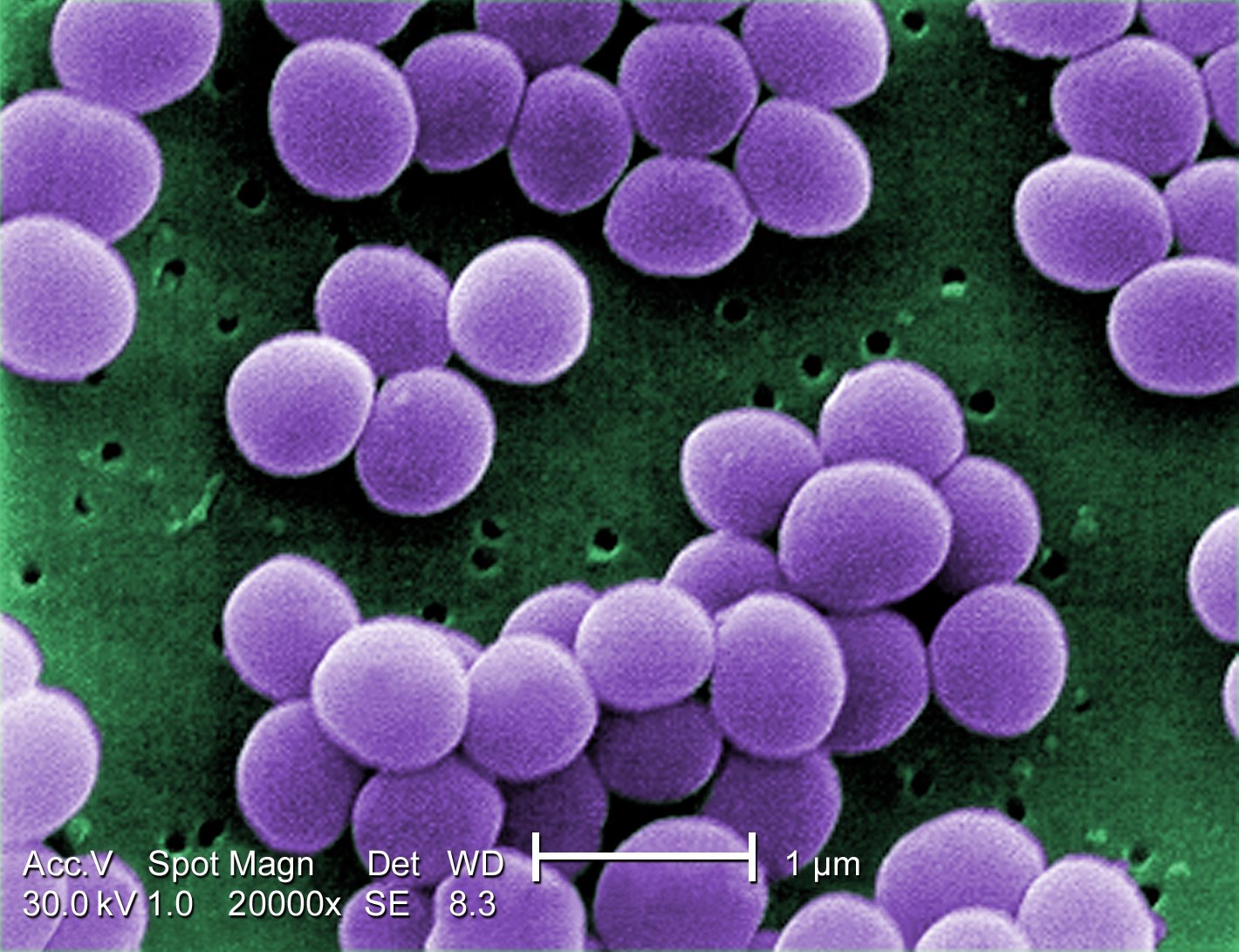 Crédito: CDC (PHIL) / Janice Haney Carr, dominio público
Crédito: CDC (PHIL) / Janice Haney Carr, dominio público
En la práctica, una sala blanca somete a los microbios a “micro-desastres” repetidos (desecación, UVC, detergentes, hambre). En vez de eliminar toda vida, el sistema puede terminar favoreciendo a los más resistentes (aunque sigan siendo pocos).
Protección planetaria: “cero microbios” no es realista, pero sí controlable
Desde el punto de vista de misión, lo importante no es que exista una cantidad absolutamente nula de vida (algo difícil de garantizar), sino mantener la carga biológica bajo límites, monitorizar y evitar escenarios en los que la contaminación comprometa ciencia o bioseguridad interplanetaria.
La NASA describe explícitamente que, para prevenir contaminación “forward” y “backward”, el hardware debe esterilizarse y evaluarse y mantenerse en entornos de sala blanca con control estricto (filtrado de aire, disciplina de personal, etc.).
La lectura razonable del estudio es doble:
- Refuerza la idea de que la limpieza y el control deben ser continuos y medidos (auditorías microbianas, secuenciación, trazabilidad).
- Ajusta expectativas: incluso con protocolos intensivos, pueden persistir microbios raros y altamente tolerantes; por tanto, la protección planetaria es un problema de gestión de riesgo, no de “purismo” absoluto.
Un giro interesante: potencial biotecnológico en microbios de sala blanca
Más allá de la preocupación por la contaminación, el estudio destaca que algunas de estas nuevas especies podrían ser útiles por su maquinaria metabólica:
- Presencia de clústeres biosintéticos asociados a ε-poly-L-lysine (un compuesto con aplicaciones en conservación de alimentos y biomedicina) en especies como Agrococcus phoenicis y Microbacterium spp.
- Dos nuevas especies de Sphingomonas con potencial para producir zeaxantina (antioxidante de interés nutricional).
- Genes para bacillibactina (captación de hierro) y otros metabolitos secundarios con interés industrial o farmacéutico, además de compuestos como alkylresorcinols en Georgenia phoenicis.
Este punto es clave: los cleanrooms son laboratorios involuntarios de selección extrema. En un ambiente donde “casi nadie” puede vivir, cualquier microbio que persiste merece atención, tanto por riesgos operativos como por oportunidades científicas.
Aclaraciones importantes
Conviene aclarar algunos puntos que podrían malinterpretarse:
- No se ha demostrado que Phoenix contaminara Marte. De hecho, el trabajo se centra en aislados de cleanroom y en su caracterización genómica, no en rastreo de contaminación en destino.
- La “capacidad de sobrevivir en el espacio” es una posibilidad razonable para microorganismos tolerantes, pero exige ensayos específicos (radiación, vacío, ciclos térmicos, etc.) para cada cepa. Algunas coberturas lo discuten como línea futura de trabajo.
- Tampoco implica que las salas blancas sean “ineficaces”: más bien muestra que la eficacia se mide en reducción, monitorización y prevención, no en eliminación total.
Referencias y lecturas recomendadas
- Artículo científico (acceso abierto, Microbiome) — “Genomic insights into novel extremotolerant bacteria isolated from the NASA Phoenix mission spacecraft assembly cleanrooms.”
- Space.com — “Not-so-clean rooms: Scientists discover 26 new microbe species in NASA spacecraft facility.”
- Live Science — “Never-before-seen ’extreme’ microbes surrounded NASA robot before it was sent to Mars 18 years ago.”
- KAUST — “Tough microbes found in NASA cleanrooms hold clues to space survival and biotech.”
- Phys.org — “Tough microbes found in NASA cleanrooms hold clues…”
- NASA Planetary Protection — Requisitos de esterilización, evaluación y uso de cleanrooms.
- NASA Brand Center — Directrices para uso de imágenes y medios.
- CDC Agency Materials — Condiciones generales de uso y atribución recomendada.


